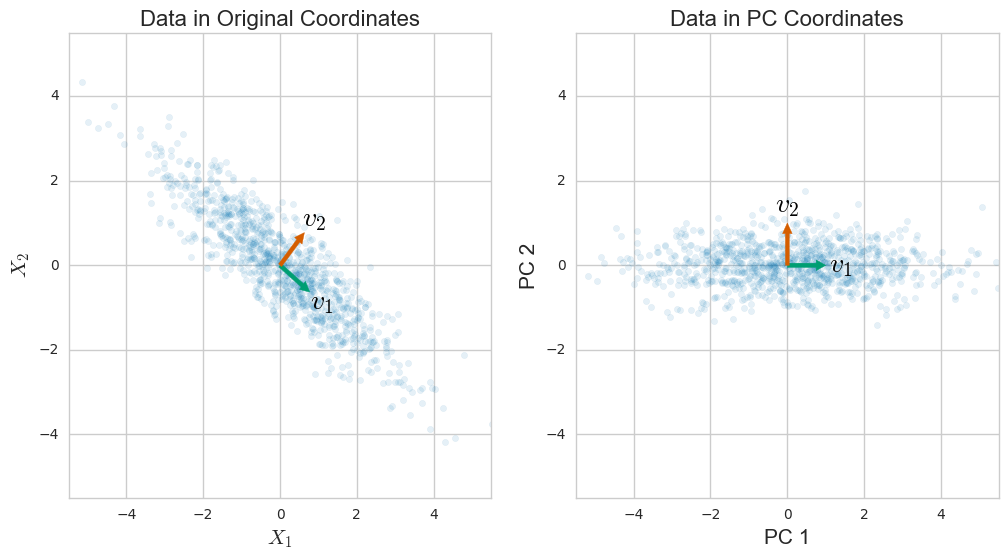
Lassen Sie mich mit PCA beginnen. Angenommen, Sie haben n Datenpunkte, die jeweils aus d Zahlen (oder Dimensionen) bestehen. Wenn Sie diese Daten zentrieren (subtrahieren Sie den mittleren Datenpunkt von jedem Datenvektor ), können Sie die Daten stapeln, um eine Matrix zu erstellenμxi
X=⎛⎝⎜⎜⎜⎜⎜⎜xT1−μTxT2−μT⋮xTn−μT⎞⎠⎟⎟⎟⎟⎟⎟.
Die Kovarianzmatrix
S=1n−1∑i=1n(xi−μ)(xi−μ)T=1n−1XTX
misst, in welchem Maße die verschiedenen Koordinaten, in denen Ihre Daten angegeben sind, voneinander abweichen. Daher ist es vielleicht nicht verwunderlich, dass PCA - mit dem die Variation Ihrer Daten erfasst werden soll - in Form der Kovarianzmatrix angegeben werden kann. Insbesondere stellt sich die Eigenwertzerlegung von als herausS
S=VΛVT=∑i=1rλivivTi,
wobei die te Hauptkomponente oder PC ist und der te Eigenwert von und auch gleich der Varianz der Daten entlang des ten PC ist. Diese Zersetzung kommt aus einem allgemeinen Satz in der linearen Algebra, und einige Arbeit nicht getan werden , haben die relatino zu PCA zu motivieren.viiλiiSi
SVD ist eine allgemeine Methode, um eine Matrix in Bezug auf ihren Spalten- und Zeilenraum zu verstehen. (Es ist eine Möglichkeit, eine Matrix in Bezug auf andere Matrizen mit einer intuitiven Beziehung zum Zeilen- und Spaltenraum neu zu schreiben.) Zum Beispiel für die Matrix Wir können die Richtungen und in der Domäne und im Bereich finden, so dassA=(1021)uivi
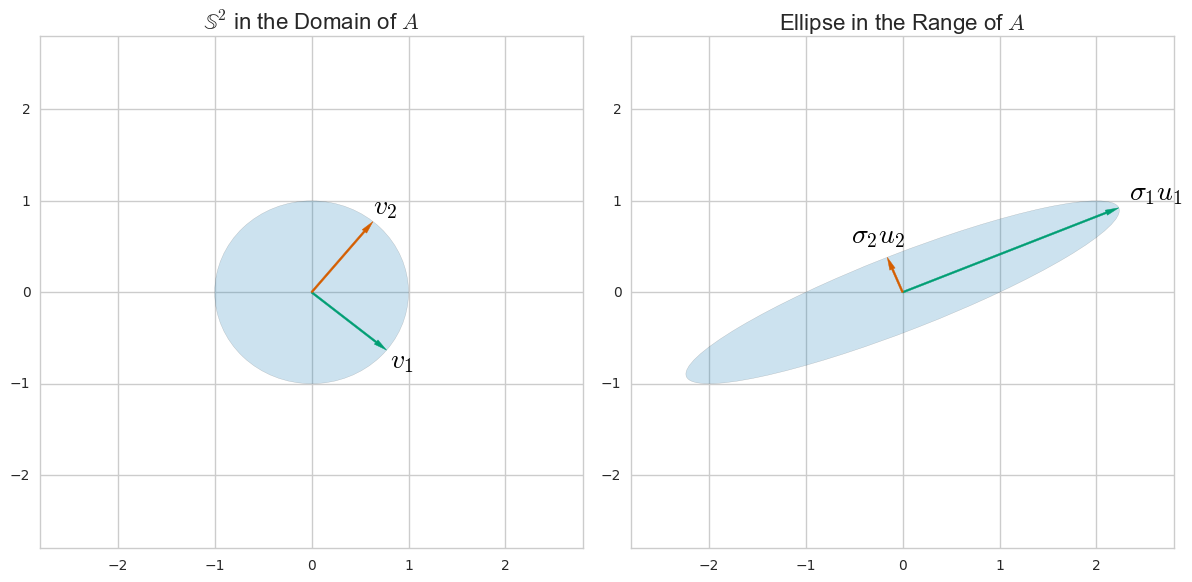
Sie können diese finden, indem Sie sich überlegen, wie als lineare Transformation eine Einheitskugel in ihrer Domäne in eine Ellipse verwandelt : Die der Ellipse richten sich nach dem und dem sind ihre Vorbilder.ASuivi
In jedem Fall lässt uns SVD für die obige Datenmatrix (setzen Sie einfach ) schreibenXA=X
X=∑i=1rσiuivTj,
wobei und orthonormale Sätze von Vektoren sind. Ein Vergleich mit der Eigenwertzerlegung von zeigt, dass die "rechten Singularvektoren" gleich den PCs sind, die "rechten Singularvektoren" sind{ v i } S v i{ui}{vi}Svi
ui=1(n−1)λi−−−−−−−−√Xvi,
und die "singulären Werte" beziehen sich auf die Datenmatrix überσi
σ2i=(n−1)λi.
Es ist eine allgemeine Tatsache, dass die rechten Singularvektoren den Spaltenraum von überspannen . In diesem speziellen Fall eine skalierte Projektion der Daten auf die Richtung der ten Hauptkomponente. Die linken singulären Vektoren überspannen im Allgemeinen den Zeilenraum von , was uns einen Satz orthonormaler Vektoren gibt, die die Daten ähnlich wie bei PCs überspannen. x u i x i v i xuiXuiXiviX
In diesem längeren Artikel gehe ich auf einige Details und Vorteile der Beziehung zwischen PCA und SVD ein .


